新規遺伝子変異の病原性予測法 MOVAを開発
2023年05月25日
概要
筋萎縮性側索硬化症(ALS:amyotrophic lateral sclerosis)(注1)は代表的な神経難病です。ALS患者さんの90%は孤発性(注2)ですが、家族性ALSの原因となりうる遺伝子変異がしばしば見出されます。これらの変異の病原性予測に、in silico(注3)解析法が使われています。ALS原因遺伝子のいくつかは、変異が特定の領域(hotspot)に集中していますが、既存のin silico解析法はそれを十分に考慮できていませんでした。
新潟大学脳研究所脳神経内科学分野の畠野雄也非常勤講師、同研究所脳神経疾患先端治療研究部門の石原智彦特任准教授、同研究所脳神経内科学分野の小野寺理教授の研究グループは、蛋白質の立体構造上の位置情報に重きを置くことで、hotspotを考慮に入れた、新しいin silicoの病原性予測法MOVA(method for evaluating the pathogenicity of missense variants using AlphaFold2)を、機械学習法を用いて開発しました。
研究成果のポイント
・蛋白質の立体構造上の位置情報に注目した新規遺伝子変異病原性予測法MOVAを開発した。
・hotspotが存在するALSの原因遺伝子で、高い予測精度が得られた。
・既存の病原性予測法と組み合わせることで、より高い予測精度が得られた。
Ⅰ.研究の背景
多くの疾患で遺伝因子が関与しています。例えば、代表的な神経難病ALSの90%は家族歴のない孤発性ですが、孤発性ALS症例でも10-30%に変異が見出されます。これらの変異の病的意義はしばしば不明です。本来であれば、培養細胞や動物実験による機能解析によって、これらの変異の病原性を検討することが必要です。しかし、見つかった多数の変異の病原性を全て培養細胞や実験動物、あるいは患者さんから得られるリソースで検討するのは、限界があります。そこで、これらの変異の病原性を予測するために、in silico解析法が広く使われています。既存のin silico解析の手法は、主に遺伝子の進化的保存とアミノ酸の類似性に基づいており、変異の遺伝子上の位置情報とその立体構造は十分に考慮されていません。
Ⅱ.研究の概要・成果
本研究グループは、遺伝子変異の立体構造上の位置情報に重きを置いた、新しいin silicoの病原性予測法MOVA(method for evaluating the pathogenicity of missense variants using AlphaFold2)を開発しました。開発には機械学習法のrandom Forestを用いました。
MOVAを12種類のALS原因遺伝子で検証し、TARDBPとFUS、SOD1、VCP、UBQLN2の5つの遺伝子でAUC(注4)0.7以上と良好な成績を示しました。既存の5つのin silico解析手法(PolyPhen-2、CADD、REVEL、EVE、AlphScore)と比較し、全体として良好な成績を示しました。
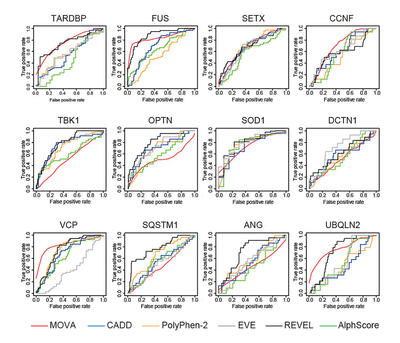
画像はHatano Y, et al.BMC bioinformatics 2023からの引用
Ⅲ.今後の展開
MOVAはALS原因遺伝子の病原性予測に有用なことが示されました。同様に他疾患の原因遺伝子にも応用が可能です。現時点では既知原因遺伝子についての予測にとどまりますが、将来的には病原性が未確定な遺伝子での変異病原性の予測法の開発も予定しています。
Ⅳ.研究成果の公表
本研究成果は、2023年5月19日、科学誌「BMC Bioinformatics」に掲載されました。
| 【論文タイトル】 |
Accuracy of a machine learning method based on structural and locational information from AlphaFold2 for predicting the pathogenicity of TARDBP and FUS gene variants in ALS |
| 【著者】 | Yuya Hatano, Tomohiko Ishihara, Osamu Onodera |
| 【doi】 | 10.1186/s12859-023-05338-5 |
用語解説
(注1)筋萎縮性側索硬化症(ALS:amyotrophic lateral sclerosis):運動神経が障害を受けることで、手足の動きや呼吸に必要な筋肉が萎縮する(やせていく)病気です。
(注2)孤発性:病気が散発的に起き、血縁者に同じ病気にかかっている人がいないことです。
(注3)in silico: in vivo(生体内)、in vitro(試験管内)に対応して、コンピューターを用いてシミュレーションで結果を予測する手法です。
(注4)AUC(Area Under the Curve): 統計学的な用語です。図中に示したグラフをROC曲線といい、その下の部分の面積をAUCと呼びます。AUCは0から1までの値をとり、値が1に近いほど判別能が高いです。





