(2018年9月3日公開)
担当:宮下哲典先生
所属:遺伝子機能解析学分野
はじめに:激増するアルツハイマー病(AD)
国際アルツハイマーリポート(文献1)によると、2015年時点の認知症患者は世界で4,680万人に達し、2050年には1億3000万人を越えると推計されている。年間あたりの新規認知症患者数は990万人と見積もられ、実に「3秒に1人」が認知症を発症する時代に突入した。今後世界の高齢化はますます加速するとみられ、それに伴う認知症の社会経済、医療経済に及ぼす影響は計り知れない。今や世界的な規模での認知症克服は危急の課題である。
認知症の半数以上はADであり、「一度発達した知的機能が脳へのアミロイドベータ(Aβ)タンパクとタウ(tau)タンパクの蓄積に伴って緩徐進行性に障害される疾患」と定義される。ADは先天的要因である個々人の遺伝情報と、後天的要因である環境要因が複雑に絡み合って発症する「ありふれた疾患」で、加齢が最大のリスクファクターである。遺伝子に関しては、変異の頻度は低いものの疾患寄与率が極めて高い遺伝子(原因遺伝子)や、逆に頻度は高いが疾患寄与率の低い遺伝子(感受性遺伝子)など(図1)、これまでに様々なものが同定されている。本コラムではADの遺伝子に焦点を絞り、我々の経験を織り交ぜながら概説する。
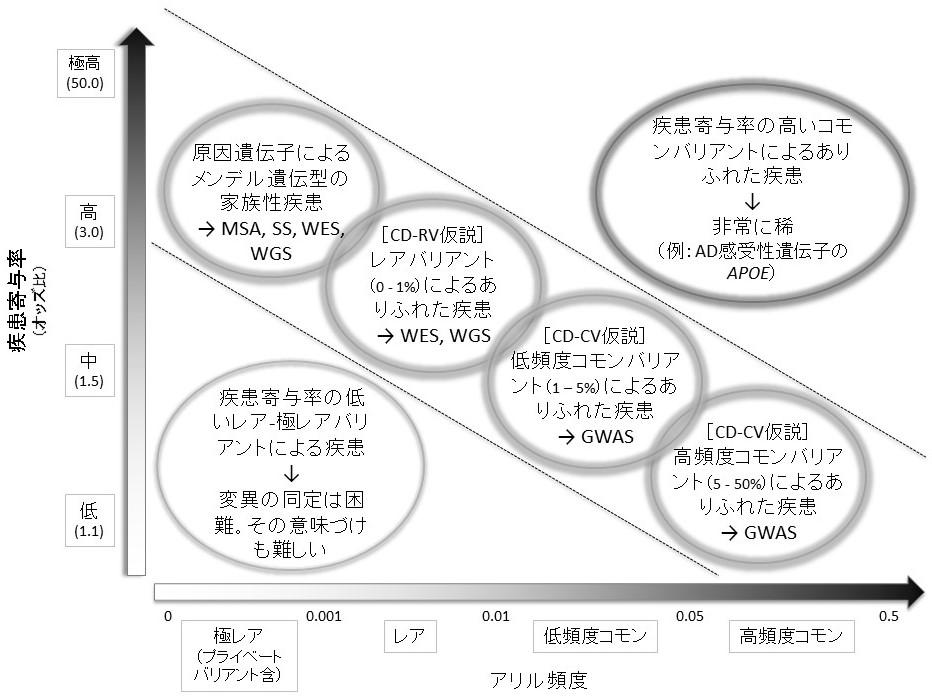
図1 | 疾患の原因・感受性遺伝子探索におけるアリル頻度と疾患寄与率の関係
文献17のボックス7と文献18の図1をもとに作成した。メンデル遺伝型の家族性疾患の原因遺伝子は、マイクロサテライトマーカーによる連鎖解析とポジショナルクローニングによって、主に1980-90年代に同定された。また、近年ではNGSを駆使した全ゲノム・エクソンシーケンシングにより、原因遺伝子が同定されている。アリルの頻度は非常に稀であるが、その疾患寄与率は非常に高い(ほぼ100%)。2000年代に入りコモンバリアントであるSNPベースのGWASが行われ、がん、糖尿病、高血圧、ADなどのありふれた疾患の感受性遺伝子が見出された。疾患に寄与するアリルの頻度は高いものの(1%以上)、その大きさは低い(オッズ比はおよそ1.5未満)。2010年代に入り、NGSによるありふれた疾患のレアバリアント探索が活発化した。民族固有の、あるいは家系固有の感受性遺伝子が相次いで報告されている。アリルの頻度は低い(1%未満)ながらもその疾患寄与率は高い(オッズ比2以上)。NGSをもってしても孤発性のありふれた疾患に関与する極レアバリアントやプライベートバリアントの同定は非常に難しく、その意味づけも困難である。一方、高頻度のコモンバリアント(5%以上)が高い疾患寄与率(オッズ比3以上)を呈してありふれた疾患に関連する例は非常に稀だが、ADの感受性遺伝子であるAPOEはそれに相当する。
原因遺伝子
1990年代に常染色体優性遺伝形式の家系を対象にしたマイクロサテライトマーカーによる連鎖解析により、若年発症型の家族性ADの原因遺伝子が特定された。1991年にAPP(21q21.3)、1995年にPSEN1(14q24.2)とPSEN2(1q42.13)がそれぞれ報告された。浸透率はほぼ100%で、親とおおよそ同じ年齢に達すると発症する。これらの遺伝子がコードするタンパクは全てAβの産生に関与することから「Aβ仮説」(文献2)が提唱され、それに基づく数多くの基礎研究、臨床研究が行われてきた。本仮説に議論の余地はあるものの病的Aβ、とりわけAβ42の代謝異常(産生増加、異常蓄積、クリアランス不全など)をAD発症の元凶として捉えることは重要である。
これまでに報告されたAPP、PSEN1、PSEN2の変異情報は我々のもの(文献3)を含め以下の3つのデータベースにアクセスすれば入手可能である。興味のある方は是非活用頂きたい。
・AD&FTDM db: Alzheimer Disease & Frontotemporal Dementia Mutation Database
・Alzforum Mutations
・JFADdb: Japanese Familial Alzheimer's Disease Database(文献3)
感受性遺伝子
APOE
APOE(19q13.32)は人種を超えた最大かつ最強のAD感受性遺伝子である。4つのエクソンから構成され、遺伝子サイズは4kb弱である。317アミノ酸(分子量36.2KDa)からなるタンパクとして合成され、N末端のシグナルペプチド(18アミノ酸)が切断された後、299アミノ酸(分子量34.2KDa)から成る成熟APOEとなる。112番目(Cys[TGC]/Arg[CGC]:rs429358)と158番目(Arg[CGC]/ Cys[TGC]:rs7412)のアミノ酸の組み合わせによって、APOE-ε2(Cys-Cys)、-ε3(Cys-Arg)、-ε4(Arg-Arg)の3種類のアイソフォームが存在し、それらに対応するアリルはそれぞれAPOE-ε2、-ε3、-ε4である。各アリルの頻度は人種差はあるもののAPOE-ε3>APOE-ε4>APOE-ε2の順になっている(文献4)。APOE-ε3r(CGC[Arg]-TGC[Cys])アリルは現人類にはほぼ存在しない(文献5)。
ADとAPOEの最初の関連は1991年のNambaら(文献6)の報告にさかのぼる。彼らはAD脳に特徴的な構造物である老人斑と神経原繊維変化に、APOEが共局在していることを免疫組織化学的に初めて示した。1993年にはRosesらのグループが家族性、及び孤発性の晩期発症型ADを対象とした分子遺伝学的解析によって、APOEとADの関連を立て続けに証明した。その後、日本人を含む様々な人種で検証され、APOE-ε3アリルに対してAPOE-ε4アリルはADの強力なリスクファクターであること、反対にAPOE-ε2アリルは防御的ファクターであることなどが明らかにされた。自験例による解析でも同様の結果が得られており、APOE-ε3*3に対するAPOE-ε4*4の粗オッズ比(95%信頼区間)は28.78(16.33 - 50.74)に達した(文献14)。
その他にもAPOEの遺伝型・アリルは認知機能、脳構造、液性バイオマーカー(Aβやtau)などの量的形質と強い相関が認められる。特にAPOE-ε4アリルの保因者はAD発症のリスクが高いことから、予防介入を試みる探索的治験が海外で展開されている(文献7)。
コモンバリアントによる探索
SNPはアリル頻度が1%以上の1塩基多型で、いわゆるコモンバリアントである(図1)。2000年代に入りこのSNPを高密度に搭載したジーンチップが開発され、疾患感受性遺伝子のゲノムワイド関連解析(GWAS)が本格化した。ADのGWASが初めて報告されたのは2007年である(文献8)。その後、欧米を中心に複数の研究グループが組織され、白人を対象とした大規模検体による解析が展開された。Lambertら(文献9)による報告はその代表で、結果を一部改変して図2と表に示す。
日本人を対象にしたGWASは2報ある(文献10,11)。我々はJapanese Genetic Consortium for Alzheimer Disease(JGSCAD)を組織し、アメリカ合衆国のAlzheimer Disease Genetic Consortium(ADGC)、及び韓国と国際共同研究を実施した(文献10)。APOEとその周辺領域に加え、SORL1(11q24.1)がADと関連することが分かった。Holstegeらの最近の報告では、APP、PSEN1、PSEN2、APOEと並んでSORL1の病的変異検索の重要性が提案されている(文献12)。注力すべき重要なAD感受性遺伝子の1つと言えよう。Hiranoら(文献11)のGWASではCNTNAP2(7q35-q36.1)が同定された。この遺伝子の発現量はADの海馬で減少しているとの報告があり、興味深い。
ADの最初のGWAS報告から11年(2018年時点)が経過した。数十を超える報告がある中、APOEを含む領域は人種を超えて、強力に、且つゲノムワイドなレベルでADと関連することが証明された。また、Lambertらの報告(文献9)に代表されるように、白人ではBIN1(2q14.3)、CLU(8p21.1)、CR1(1q32.2)、PICALM (11q14.2)は確実にADと相関し、再現性が認められる。従って、これら4遺伝子は白人ではAPOEに次ぐAD感受性遺伝子と考えられる。一方、日本人のGWASではSORL1(文献10)やCNTNAP2(文献11)が見出されたが、これらをきちんと確定するには、日本人を含むアジア人の複数の大規模検体によるメタ解析が必要であろう。
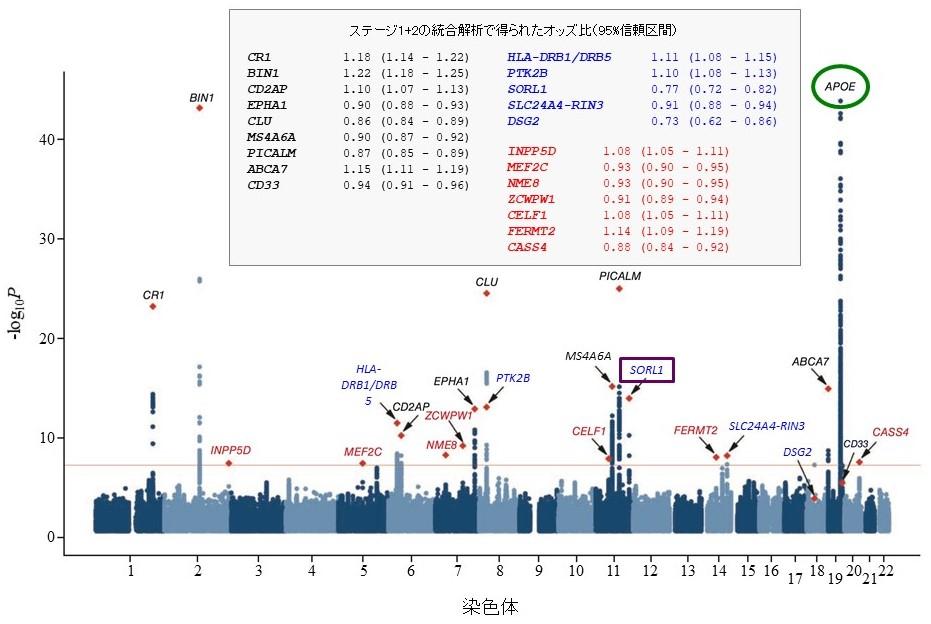
図2 | 欧米人による大規模メタGWASによって認められたAD感受性遺伝子
文献9の図1を改変した。欧米人のAD17,008例と対照37,154例を用いたステージ1の解析のマンハッタンプロット。既報のGWASで同定されていた遺伝子は黒字(CR1、BIN1、CD2AP、EPHA1、CLU、MS4A6A、PICALM、ABCA7、APOE、CD33)、新たに見出された遺伝子を青字(ステージ1の新規遺伝子:HLA-DRB5-DRB1、PTK2B、SORL1、SLC24A4-RIN3、DSG2)と赤字(ステージ1+2の新規遺伝子:INPP5D、MEF2C、NME8、ZCWPW1、CELF1、FERMT2、CASS4)でそれぞれ示す。最強のAD感受性遺伝子APOEは緑色の丸で囲んだ。APOE周辺領域では複数のSNPが強力に相関することが分かる。SORL1は我々のGWAS(文献10)でも同定されたので紫色の四角で囲み強調した。
レアバリアントによる探索
SNPベースのGWASによって同定されたAD感受性遺伝子のオッズ比は、APOEを除き、およそ1.5未満と非常に小さい(図2-3、表)。また、関連を示すほとんどのSNPは遺伝子間やイントロンに位置しているため、遺伝子機能に与える影響はさほど大きくないと推定される。APOEに匹敵する強力なAD感受性遺伝子が4〜7個ほど存在しているとする報告があることから、そのような遺伝子をあぶり出すための新たな技術革新、解析手法が待望されていた。
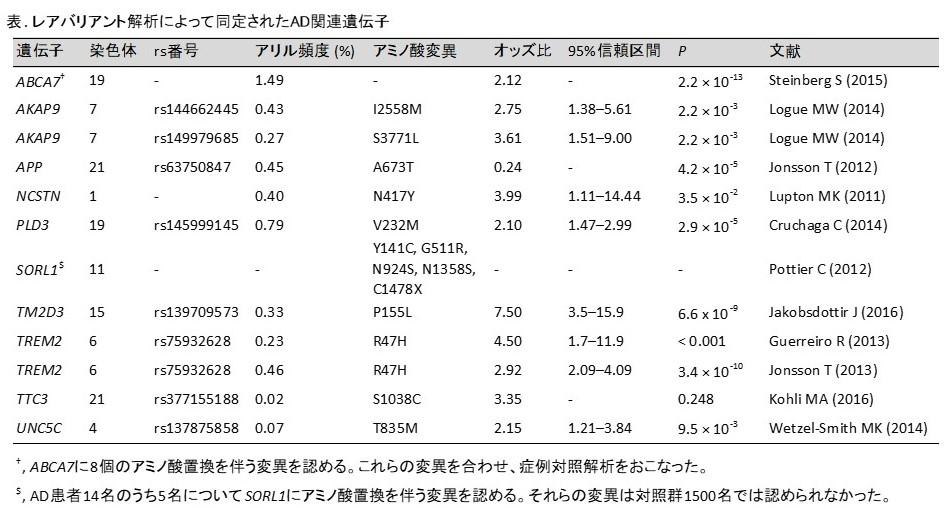
2010年代に入り、大規模並列型DNA塩基配列解析装置である次世代シーケンサー(NGS)の活用が活発化したことで、ブレークスルーが起きた。この装置により全ゲノムを1塩基の解像度で同時に、且つ網羅的に読み取ることが可能となり、アリル頻度1%未満のレアバリアントが解析対象に加わった。レアバリアントがアミノ酸置換を伴う場合、遺伝子機能に直接影響を及ぼすことが想定される。従って、疾患との関連が疑われた場合、培養細胞や動物モデルを用いた機能解析を容易に行えるという利点がある。このレアバリアントに着目したNGSベースのアプローチにより、APOEに匹敵するオッズ比を示す遺伝子が同定されている。それらを表にまとめた。ちなみにSimsら(文献13)は大規模検体を対象にジーンチップベースのレアバリアント解析を行い、AD感受性遺伝子の同定に成功している。
今後も遺伝性の濃厚な家系や大規模検体を対象にしたレアバリアント解析によって、新規のAD感受性遺伝子が発見されると予想される。しかし、人種が異なると再現できない場合があり、実際に我々も欧米人で報告されたTREM2(6p21.1)(文献14)やAPP(未発表データ)のレアバリアントが日本人では全く存在しない、あるいは相関しないことを経験した。これは集団固有の、さらには家系固有のAD感受性遺伝子が存在していることを示唆している。日本人のADに寄与するレアバリアントを同定するためには、我々日本人の検体を用いた地道な解析努力が必要である。
おわりに:遺伝子によるAD発症リスク予測への期待
およそ30年をかけADの原因や感受性に関与する遺伝子が明らかにされてきた(図2-3、表)。現在はNGSを用いたレアバリアント解析が主流となっており、民族固有、家系固有の遺伝子(変異)がさらに同定されるものと予想される。今後ADの予防介入を視野に入れた取り組みとして、発見された遺伝子をどのように有効活用するか?遺伝学的にリスクの高い人を事前に選抜し、生活習慣(運動、食事、知的活動など)の変化を適切に促したり、薬剤を投与したりすることで、ADの発症や進行を遅らせることができるかもしれない。欧米では実際にこれまでに得られた膨大なSNP情報を用いてポリジェニックスコア(polygenic score)(文献15)、あるいはポリジェニックハザードスコア(polygenic hazard score)(文献16)を算出し、ADの先制医療に活用しようとする取り組みが模索されている。日本人のADに最適な予防法と個別化医療を確立するために、我が国の遺伝的要因に関するデータを独自に積極的に収集し、蓄積し、共有し、活用することが今後必要である。真摯に向き合っていきたい。
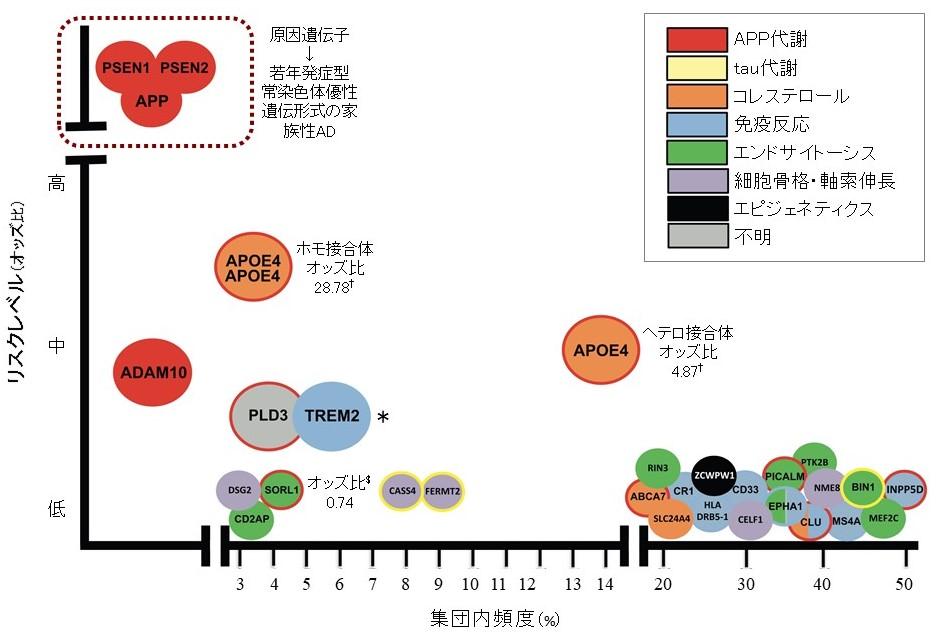
図3 | ADの原因・感受性遺伝子
文献19の図1を改変した。欧米人による遺伝学的解析で見出されたADの原因・感受性遺伝子の集団内頻度とリスクレベルとの関係を示す。各遺伝子は機能に応じて色分けされている。†, TREM2の解析(文献14)に使用した検体(臨床+病理検体:疾患群2,190例、対照群2,498例)で算出した。APOE-ε3*3に対するAPOE-ε4*4(28.28)とAPOE-ε3*4(4.87)のオッズ比を示す。*, TREM2は日本人を含む東アジア人のADとは相関しない。$, 我々の報告したSORL1のSNP rs3781834のオッズ比(0.74)を示す(文献10)。
補足:本コラムは文献20を改変しました。より詳細な内容は文献20をご覧下さい。