(2018年5月7日公開)
担当:阿部学先生
所属:モデル動物開発分野
はじめに
前回のコラムでは、心の場である脳を構成する神経の活動は化学的な反応によって生じていることと、脳機能を分子レベルから理解するにはノックアウトマウス(特定の分子が働かなくなるよう遺伝子を改変されたマウス)が有用なツールの一つであることについて紹介しました。今回は「記憶・学習に関与する分子群」について述べる予定でしたが、近年この遺伝子改変技術に関して革命的とも言えるゲノム編集技術の発展があったことを踏まえ、脳研究の話題からは少し離れますが、遺伝子工学と発生工学の最近の動向などについて紹介したいと思います。
発生工学の進歩とノックアウトマウス

(左上)当研究室で独自に樹立したC57BL/6N系マウスES細胞(RENKA細胞)。
(左下)マイクロインジェクション法によるキメラマウス作製。マウス8細胞胚中に数個のES細胞を注入することでキメラマウスが作製できる。
(右)作製されたキメラマウス。黒色に近いほどES細胞に由来する細胞の比率が高い。右端のマウスはほぼ全身の細胞がES細胞由来だと考えられる。
相同組換えを利用したジーンターゲティング法とマウスES細胞(胚性幹細胞)からのキメラマウス作製技術の組み合わせにより(図1)、マウスにおいてはほぼ制約無く任意の内在遺伝子の欠損を可能とする遺伝子ノックアウト法が確立されました。このノックアウトマウスと健常なマウスの表現型を比較することで標的遺伝子の生理機能を明らかにするのですが、この手法は言い換えると「遺伝子という生命の設計図からのアプローチ」であり、「分子と個体をつなぐ研究方法」となるため脳研究においても非常に有効な解析手法となります。しかし標的遺伝子によっては通常のノックアウト法では変異個体が致死となることもあれば、類縁分子による機能的代償が生じることもあり、正確な遺伝子の機能評価ができない場合があります。
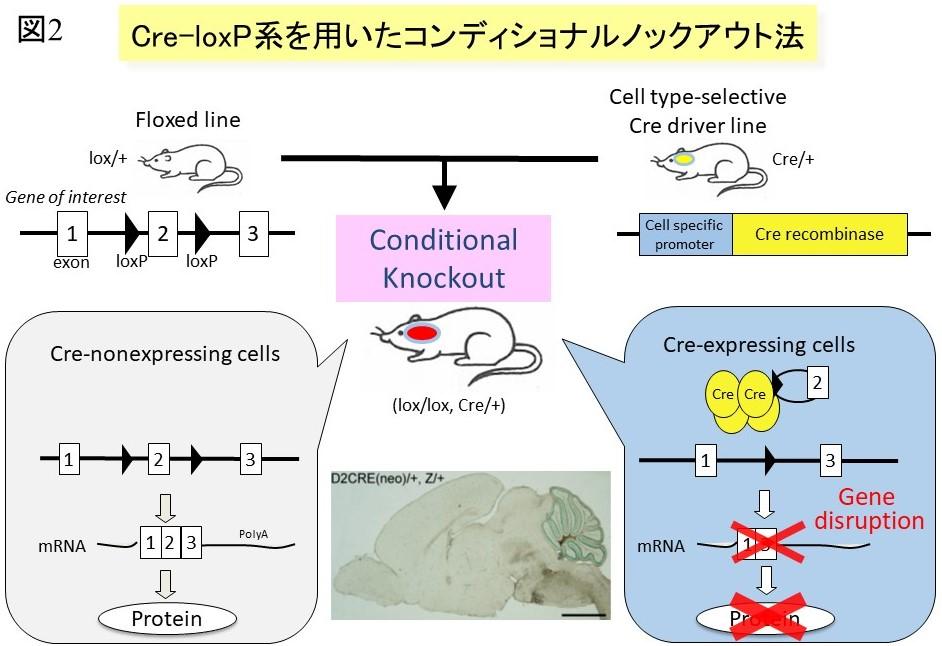
これらの欠点を回避するため開発された手法が「コンディショナル(条件的)ノックアウト法」です。コンディショナルノックアウト法で最もよく適用されているシステムは、分子生物学実験でもよく用いられているP1ファージ由来のCre-loxP組換え系であり、DNA組換え酵素であるCreタンパクがDNA配列に存在する2カ所のloxP配列を組換えてその配列間を切り出すという原理に基づいています。遺伝子改変マウスに適用する場合、一般的には生体内でCreタンパクの発現部位と発現時期が予め調べられているトランスジェニックマウス(Cre発現マウス、ドライバーマウス)と、標的遺伝子の一部または全体を挟むようにジーンターゲティング法でゲノムDNAの2カ所にloxP配列を挿入されたマウス(floxed変異マウス)を交配させます。そして得られたダブルトランスジェニックマウスがCre活性に対応した時空間特異的な遺伝子ノックアウトマウスとなり(図2)、解析に用いられます。例えば全身で欠損した場合には致死となり成体で解析できない遺伝子であっても、神経細胞でだけCre活性を持つドライバーマウスを用いることで致死性を免れて、神経系での遺伝子の機能を評価できる可能性があるということです。Cre発現マウス、floxed変異マウスとも充分に使用実績があり研究目的に合致する系統ならば、作製者から直接、または理研バイオリソースセンター(http://mus.brc.riken.jp/ja/)等の分与機関から入手するのが一般的です。しかし最適な系統が常に入手可能であるとは限らず、その場合は研究者個人がデザインして遺伝子改変マウスの作製を試みることになりますが、遺伝子改変マウス作製には発生工学、遺伝子工学を中心とする専門的な設備とスキルが必要となるため、未経験者が独力で作製するというのはほぼ不可能に近く、特に脳研究において重要な位置を占める行動学的解析においては使用するマウスの系統の選択も極めて重要であるため(Mishina & Sakimura, 2007)、いずれのマウス作製についても経験豊富な研究グループに事前に相談すべきです。
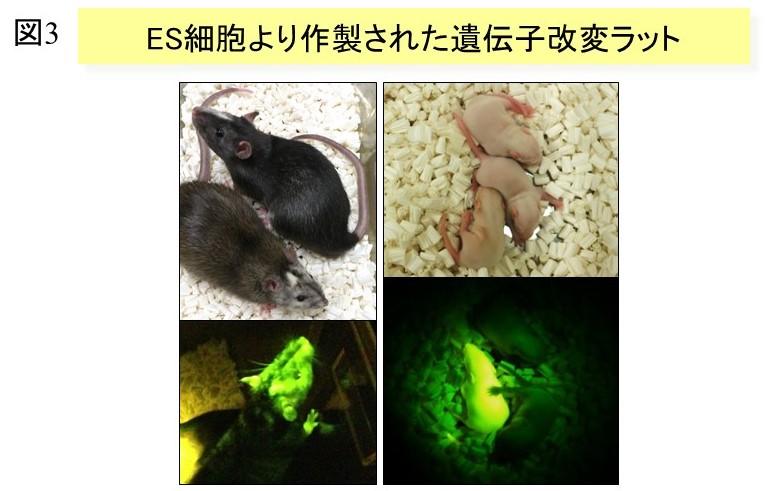
(左)当研究室で独自に樹立したES細胞より作製されたキメララット。ES細胞に導入されたVenus遺伝子の蛍光が体の一部で発現している。
(右)キメララット由来の産仔。3匹中の1匹でキメララットより生殖系列伝達したVenus遺伝子が全身で発現している。
ラットについても2008年に生殖系列伝達するES細胞が樹立されましたが(Buehr et al., 2008; Li et al., 2008)、それでも伝達効率に難があるのか実際の遺伝子改変ラットの報告数は決して多いとは言えず、ほとんど全ての哺乳動物においては未だに生殖系列伝達するES細胞が樹立されていない状況です。我々のグループでも独自にラットからES細胞を樹立し遺伝子改変ラットを作製することに成功しましたが(図3)、やはり作製の効率化は重要な課題となっています。しかし近年の幹細胞研究の進展により、他の多くの動物種からの生殖系列伝達する幹細胞の樹立に道が拓かれつつあると思われます。
ゲノム編集技術
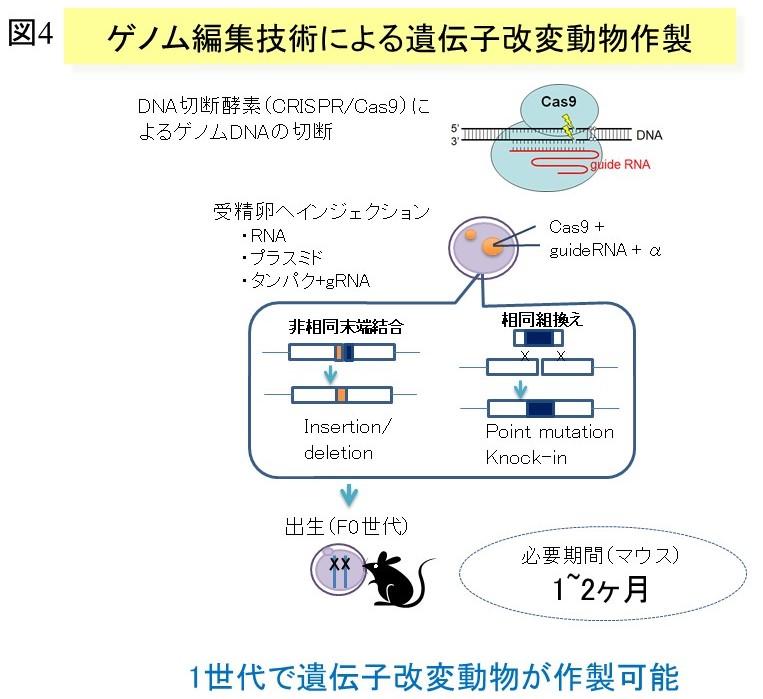
一方、ES細胞を用いない遺伝子改変技術として代表的なものはゲノム編集技術です。任意のDNA配列を特異的に認識して切断するよう設計されたDNA切断酵素を細胞へ導入することにより、ゲノムDNAの切断と修復を繰り返すうちに変異を引き起こすという原理に基づいており、受精卵で直接遺伝子改変が可能であるため、ES細胞を介した遺伝子改変マウス作製と比較して作製速度においては極めて有利な技術です。さらに原理的に全生物に適用できる可能があることも重要な点です。ゲノム編集技術の中でも、その実験の容易さと高い変異導入効率から爆発的な勢いで拡がっているのがCRISPR/Casシステムであり(Cong et al., 2013; Mali et al., 2013)、標的とするゲノムDNA配列に対応した20塩基対の配列を含む短いguide RNAとDNA切断酵素であるCas9(タンパク、mRNA、または発現プラスミド)と共に受精卵へ導入するだけでノックアウト動物が作製可能です(Mashiko et al., 2013; Wang et al., 2013)。点変異の導入やノックイン法の効率化も進み(Aida et al., 2015; Yoshimi et al., 2016)、すでに様々な遺伝子改変動物作製法として実用段階にあります(図4)。さらに、熟練を要する発生工学的技術すら不要となるようなエレクトロポーレーション法を用いた簡便な手法も確立されています(Gurumurthy et al., 2016; Kaneko & Mashimo, 2015)。当然、前述のコンディショナルノックアウト法にも適用が可能であるため、これらのシステムは将来的に遺伝子改変動物作製技術の中核になると予想されています。
終わりに
ゲノム編集技術はその汎用性の高さから、遺伝子改変動物作製のみならず生命科学研究全体、また医療や産業にまでも多大な影響を及ぼしています。日本の神経科学界ではゲノム編集技術により霊長類であるコモンマーモセットの遺伝子改変を試みており成果を挙げつつあります。今後の遺伝子改変動物を用いた脳研究は、リソースとして蓄積された樹立済みの系統を有効利用しつつ、さらに最新のゲノム編集技術を駆使して研究者独自の視点で作製されたツールが有機的に関連し、発展してゆくであろうと期待されています。





